Christopher Johnson
Laboratorium Badań Medycznych Biologii Molekularnej, Cambridge, UK
Sophia Kenrick
Dział Badań i Rozwoju Wyatt Technology (USA)
strony wersji drukowanej: 30-32
strony wersji drukowanej: 30-32
Sprzężenie chromatografii żelowej z detekcją wielokątowego rozpraszania światła dostarcza wysokiej jakości danych i ułatwia naukowcom zrozumienie ludzkiego zdrowia oraz stanów chorobowych.

Rysunek 1: Optilab UT-rEX Wyatt’a jest detektorem refraktometrycznym specjalnie zaprojektowanym do systemów UHPLC wykorzystujących kolumny wypełnione małym ziarnem.
Detekcja rozpraszania światła pod wieloma kątami jednocześnie (Multi-angle light scattering, MALS) stała się niezbędnym narzędziem charakterystyki biofizycznej do wyznaczania mas cząsteczkowych bez odnoszenia się do standardów mas cząsteczkowych. Często jest stosowana w sprzężeniu z systemami rozdziałowymi takimi jak: frakcjonowanie w polu sił przepływu (FFFF - Flow Field Flow Fractionation) lub chromatografia żelowa (SEC - Size Exclusion Chromatography). MALS służy do wyznaczania absolutnych mas cząsteczkowych, wielkości i polidyspersyjności próbek biologicznych oraz innych makromolekuł. Za pomocą dynamicznego rozpraszania światła online, ten sam eksperyment może dodatkowo dostarczyć informacji o wielkości hydrodynamicznej molekuł. Ponieważ SEC w sprzężeniu z detekcją MALS mierzy absolutne masy cząsteczkowe makromolekuł wymywanych z kolumny chromatograficznej niezależnie od ich objętości elucji, metoda ta pozwala na dokładne zmierzenie mas cząsteczkowych (Mw) lepkich lub wydłużonych białek, które wymywane są zbyt późno lub zbyt wcześnie ze złoża kolumny chromatograficznej. Ponadto MALS może być stosowany w trybie wsadowym (batch) aby zmierzyć specyficzne lub niespecyficzne oddziaływania międzybiałkowe. SEC-MALS ma wiele zastosowań, wliczając szybkie i dokładne wyznaczanie mas cząsteczkowych w roztworze, wykrywanie obecności agregatów, określanie stechiometrii kompleksów białkowych i wyznaczanie stopnia koniugacji polipeptydów (np. glikozylacji, PEGylacji oraz kompleksów białko-detergent).

W laboratorium Biologii Molekularnej przy Medical Research Council (MRC) naukowcy pracują nad lepszym zrozumieniem procesów w skali molekularnej, których znajomość jest potrzebna, aby rozwiązać kluczowe problemy ludzkiego zdrowia. Badania rozciągają się od analizy strukturalnej poszczególnych makromolekuł, ich wzajemnych oddziaływań, a także na funkcjonalne systemy sub-komórkowe, komórki oraz układy komórek w całych organizmach. Ten instytut badawczy zaadoptował technikę instrumentalną rozpraszania światła MALS jako część swojego założenia wyposażenia laboratoriów biofizycznych w najbardziej zaawansowane narzędzia, z których pomocą można uzyskiwać wiedzę niezbędną do pokonywania określonych problemów dotyczących ludzkiego zdrowia i chorób.
W typowej aplikacji badawczej poddano ekspresji i oczyszczono białko rekombinowane a następnie oszacowano jego masę cząsteczkową w roztworze. Wyznaczona za pomocą analitycznego SEC masa cząsteczkowa tego białka znajdowała się pomiędzy 200 a 700 kDa (Rysunek 2). Jednakże spodziewana w oparciu o sekwencję polipeptydu masa cząsteczkowa wynosi tylko 46 kDa. W czasie frakcjonowania na konwencjonalnych kolumnach rozdzielających, białko wyłania się w postaci dwóch pików (Rysunek 2B, czerwony), znacznie większych niż można by oczekiwać w porównaniu do wzorców białek rozdzielonych na tej samej kolumnie i systemie (Rysunek 2B, czarny).
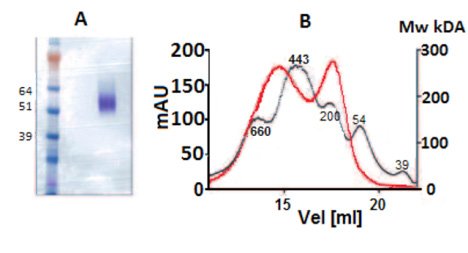
Rysunek 2: A) Białko rekombinowane migruje w pobliżu spodziewanej masy cząsteczkowej monomeru (46 kDa) w warunkach natywnego, nieredukującego SDS-PAGE. B) Chromatogram analitycznego SEC białka (czerwony) nałożony ze standardami mas cząsteczkowych (czarny).
W elektroforezie żelowej białko migruje w pobliżu spodziewanej masy cząsteczkowej. Agregacja białka indukowana tworzeniem mostka disiarczkowego została wykluczona, ponieważ w trakcie elektroforezy w warunkach nieredukujących nadal występuje monomer o prawidłowej masie (Rysunek 2A). Analiza SEC-MALS materiału z drugiego piku wykazuje, że masa cząsteczkowa jest z pewnością znacznie niższa od przewidywanej przez analityczny SEC. Zmierzona masa 60 kDa jest zbliżona do monomeru białka i zgodna z charakterystyką SDS-PAGE (Rysunek 3, zielony ślad).
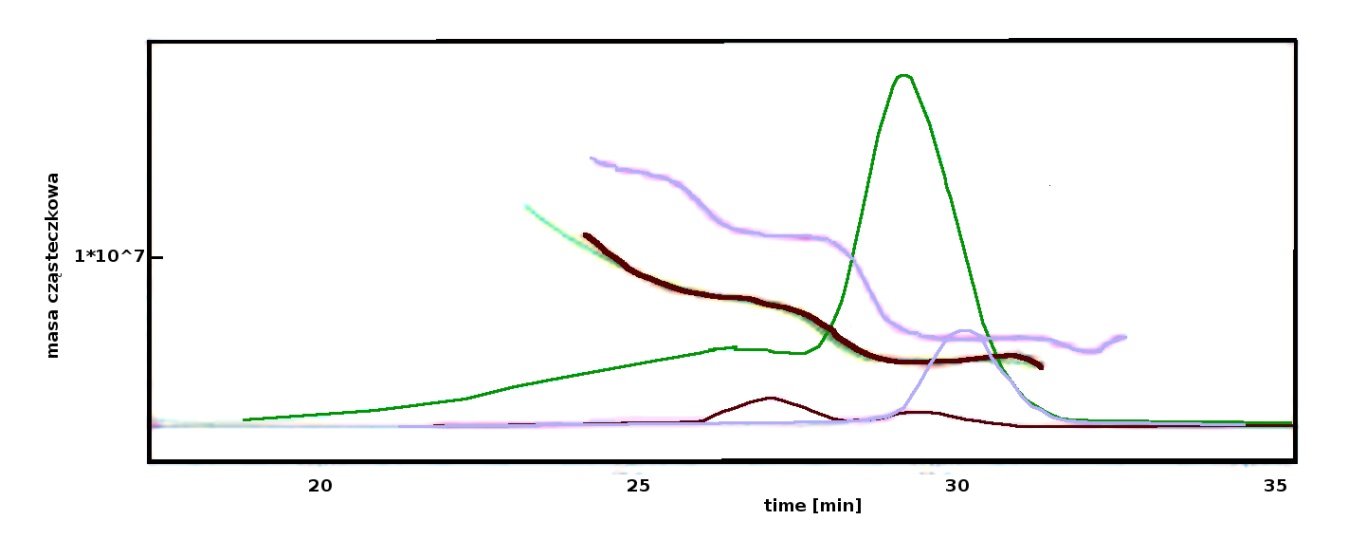
Rysunek 3: dane z analizy SEC-MALS rekombinowanego białka przy dwóch różnych nastrzykniętych stężeniach (zielony i brązowy) nałożone z danymi SEC-MALS dla albuminy osocza wołowego (BSA, fioletowy). Sygnały detektora refraktometrycznego i masy cząsteczkowe są podane dla każdego śladu.
Nieznacznie zawyżona masa (60 kDa vs. 46 kDa) może być spowodowana obecnością niewielkiej ilości cząsteczek wyższego rzędu w równowadze z monomerem. Jednakże ta ewentualność została wykluczona przez powtórzenie eksperymentu SEC-MALS przy 20-krotnie niższym stężeniu, co dało identyczne profile chromatograficzne i masy cząsteczkowe (Rysunek 3, zielone i brązowe ślady). Co ciekawe, białko to zostało poddane ekspresji w wektorach komórek eukariotycznych, a analiza sekwencji sugeruje, że może być kilka potencjalnych miejsc glikozylacji.
Ponowna analiza wyników SEC-MALS z zastosowaniem analizy koniugatów białkowych sugeruje masę cząsteczkową części białkowej 45 kDa i 10-15 kDa dla części polisacharydowej (Rysunek 4).
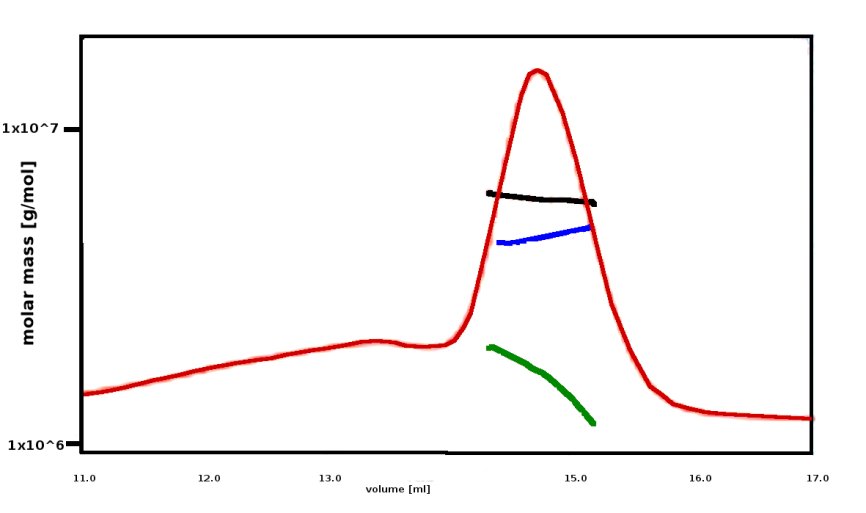
Rysunek 4: Analiza koniugatów białkowych rekombinowanego białka pokazuje, że całkowita masę cząsteczkowa 60 kDa (czarny) składa się z części białkowej ok. 45 kDa (niebieski) oraz modyfikacji polisacharydowej 10-15 kDa.
Tak obliczona wartość masy cząsteczkowej białka jest bliska masie spodziewanej z sekwencji białka a odpowiednio składowa glikozylowa jest zgodna z przewidywaną całkowitą liczbą miejsc glikozylacji oraz ich typową modyfikacją w tej linii komórkowej. W tych warunkach w roztworze białko jest zatem glikozylowanym monomerem. Jego wczesne wymywanie w SEC w porównaniu z globularnymi standardami może być wyjaśnione zmianą w jego właściwościach hydrodynamicznych spowodowaną glikozylacją. Sprawdzono to przeprowadzając jednocześnie z MALS pomiar DLS online, co pozwoliło na pomiar promienia hydrodynamicznego białka przy jednoczesnym pomiarze masy cząsteczkowej.
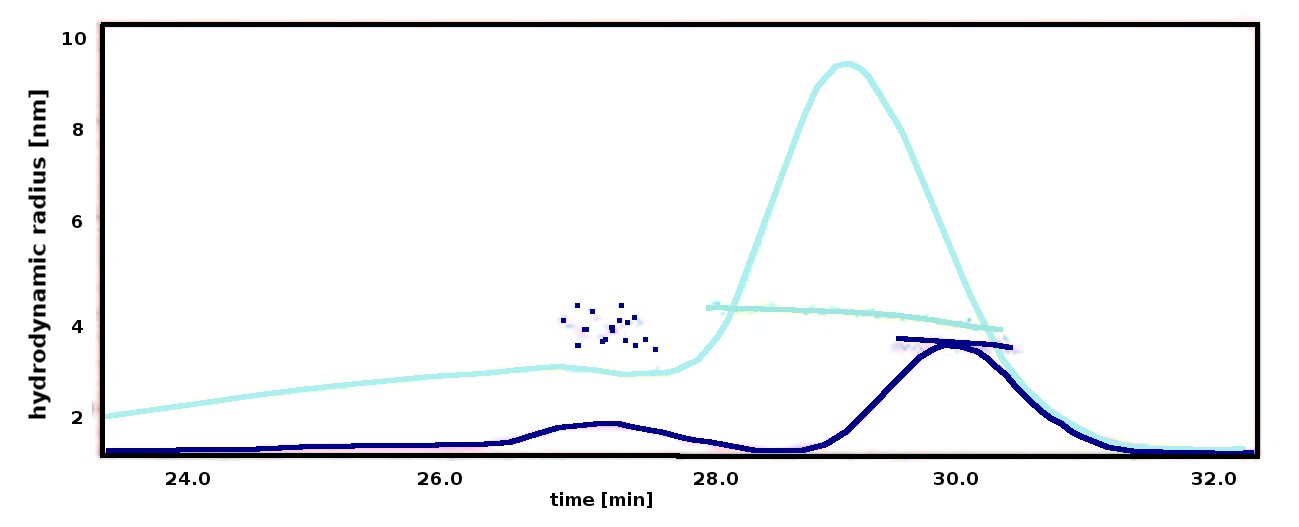
Rysunek 5: Promień hydrodynamiczny zmierzony za pomocą dynamicznego rozpraszania światła dla białka rekombinowanego (jasnoniebieski) i BSA (granatowy); białko o większej masie, ale o mniejszym promieniu wymywane jest później.
Dane DLS wskazują, że promień hydrodynamiczny tego białka wynosi 4 nm (Rysunek 5; jasnoniebieski). Białko jest zatem większe niż możnaby się spodziewać dla ,,typowego” globularnego lub niemodyfikowanego białka o takiej samej masie cząsteczkowej. Przykładowo, promień hydrodynamiczny białka monomeru osocza wołowego (BSA) o masie 66 kDa ma tylko 3,4 nm. (Rysunek 5, granatowy - główny pik ok. 30 min.). Ponieważ rozdzielanie w SEC następuje za względu na wielkość hydrodynamiczną, to wyjaśnia dlaczego cząsteczka o większym promieniu hydrodynamicznym wymywana jest wcześniej niż cząsteczka o mniejszym promieniu, pomimo że masa cząsteczkowa BSA jest większa niż masa cząsteczkowa badanego rekombinowanego białka.
Zdając sobie sprawę z korzyści jakie daje MALS w laboratorium MRC używano takiej detekcji jako jednej z części badań końcowych. Dzięki temu poprzednie koncepcje naukowe odporności na choroby wirusowe jak: przeziębienie, AIDS, zimowe wymioty i wirusowe zakażenia przewodu pokarmowego uległy znaczącym modyfikacjom. Wirusy wywołujące takie choroby są najtrudniejsze w zwalczaniu i łatwo wymykają się rozpoznaniu przez układ odpornościowy poprzez zmienianie swoich wzorców molekulanych.
Początkowo sądzono, że przeciwciała zapewniają wyłącznie zewnątrzkomórkową ochronę i są bezsilne, gdy tylko wirus dostanie się do cytozolu komórki. Z użyciem SEC-MALS udało się zidentyfikować pozytywne oddziaływanie pomiędzy cytozolowym receptorem Immunoglobuliny G (IgG), białkiem zawierającym trzyczęściowy motyw 21 (TRIM21) oraz IgG. Naukowcy odkryli, że TRIM21 łączy się z przeciwciałami z większym powinowactwem niż jakikolwiek inny receptor IgG w ludzkim ciele, a raz aktywowany, zaczyna gwałtownie degradować wiriony w cytozolu, neutralizując infekcję.
Tradycyjnie SEC lub FFF pozwalały na oszacowanie masy cząsteczkowej w oparciu na porównanie czasu elucji składnika względem wzorców zewnętrznych (SEC) lub za pomocą obliczeń w oparciu o teoretyczny współczynnik dyfuzji (FFF). Masy cząsteczkowe uzyskane z tych semi-empirycznych podejść są często błędne, zwłaszcza gdy badana próbka ma inną konformację niż standard odniesienia lub model. Dodanie wielokątowego rozpraszania światła (MALS) do systemu frakcjonującego pozwala na dokładny pomiar mas cząsteczkowych, bez odnoszenia się do standardów lub założeń odnośnie dyfuzji cząsteczkowej. Dodatkowo DLS online rozszerza możliwości analityczne, dostarczając informacji o wielkości hydrodynamicznej, która może wpływać na czas elucji.
SEC-MALS, FFF-MALS DLS online dobrze się sprawdziły w laboratorium MRC, dostarczając naukowcom wysokiej jakości danych i pozwalając na poczynienie znaczących postępów w zrozumieniu różnych procesów biofizycznych, wliczając w to mechanizmy chorób wirusowych i odporności. Techniki rozpraszania światła dostarczają także świetnego narzędzia badawczego do szczegółowej charakterystyki zjawiska agregacji w formulacjach białkowych, wnosząc w ten sposób wkład w ochronę ludzkiego zdrowia i w zwalczanie chorób za pomocą nowych biofarmaceutyków.
Tłumaczenie: Jacek Achrem-Achremowicz
Więcej informacji o technikach rozpraszania światła można uzyskać na stronie:
www.wyatt.com





