Krzysztof Brzozowski
Uniwersytet GdańskiBiałka to grupa biopolimerów, która od kilku lat przykuwa uwagę wielu grup badawczych. Są one bardzo często modyfikowane poprzez glikozylację, fosforylację czy acylację. Różnorodność tych modyfikacji warunkuje ich aktywność fizjologiczną, regulatorową czy metaboliczną.

Z uwagi na szeroki zakres właściwości biologicznych białek, kluczowe jest zrozumienie zależności aktywność-struktura. Poznanie budowy przestrzennej całej cząsteczki bądź jej fragmentów odpowiedzialnych za aktywność to wyzwanie dzisiejszej biochemii strukturalnej. Znajomość szczegółów strukturalnych pozwala na zrozumienie mechanizmu działania oraz dynamicznego zachowania dużych cząsteczek pod wpływem różnych warunków.
Jednym z klasycznych przykładów dobierania warunków rozdziału jest optymalizacja pH, która to wartość jest związana z tzw. punktem izoelektrycznym białka. Ma to z kolei wpływ na formę, w której cząsteczka występuje (kationowa, anionowa lub jon obojnaczy). Rysunek 1 pokazuje w jaki sposób pI i pH są ze sobą związane.
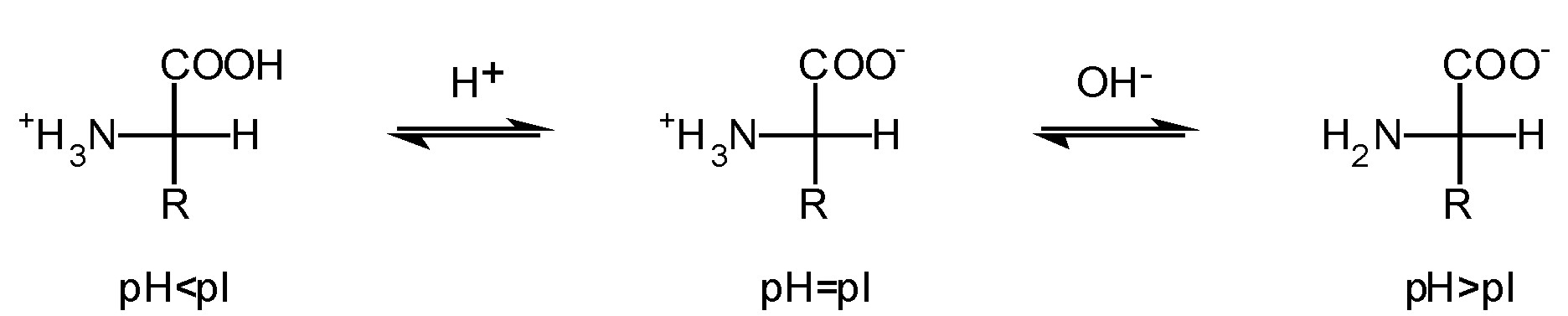
Rysunek 1. Zależność pomiędzy pH a pI aminokwasu
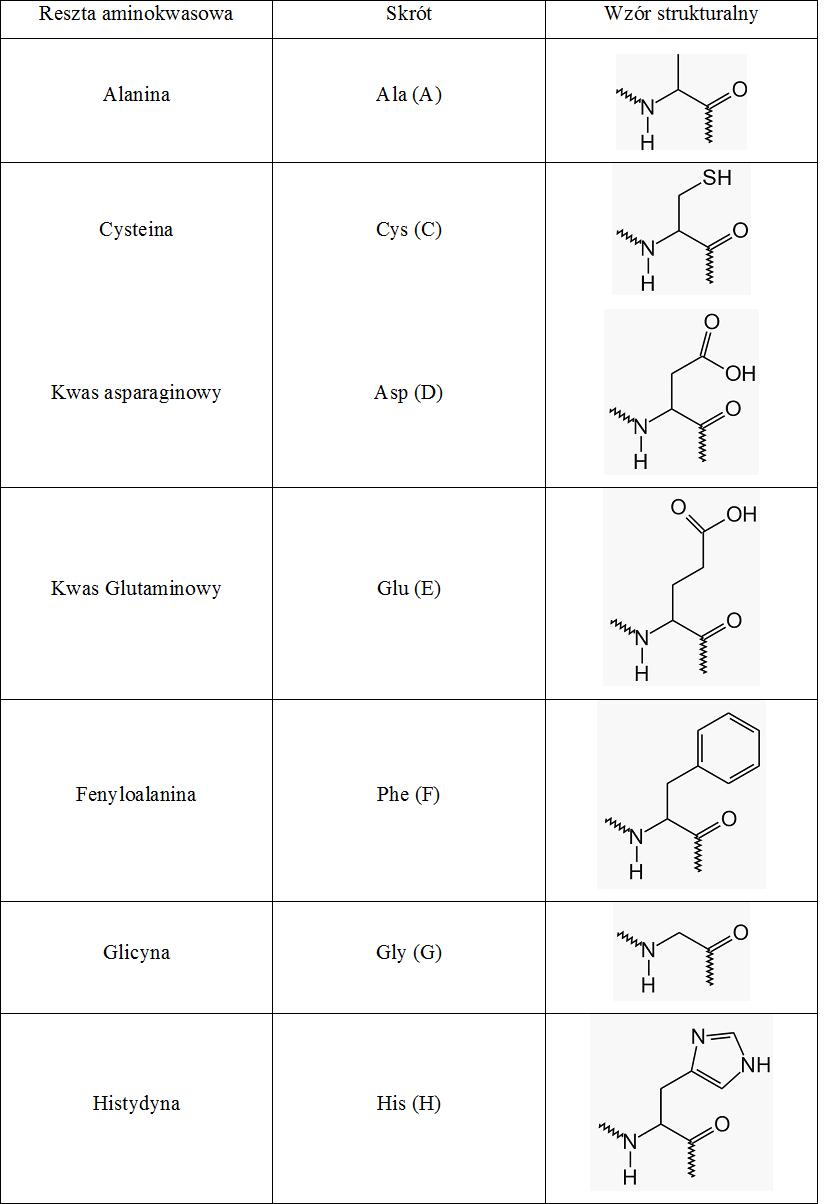
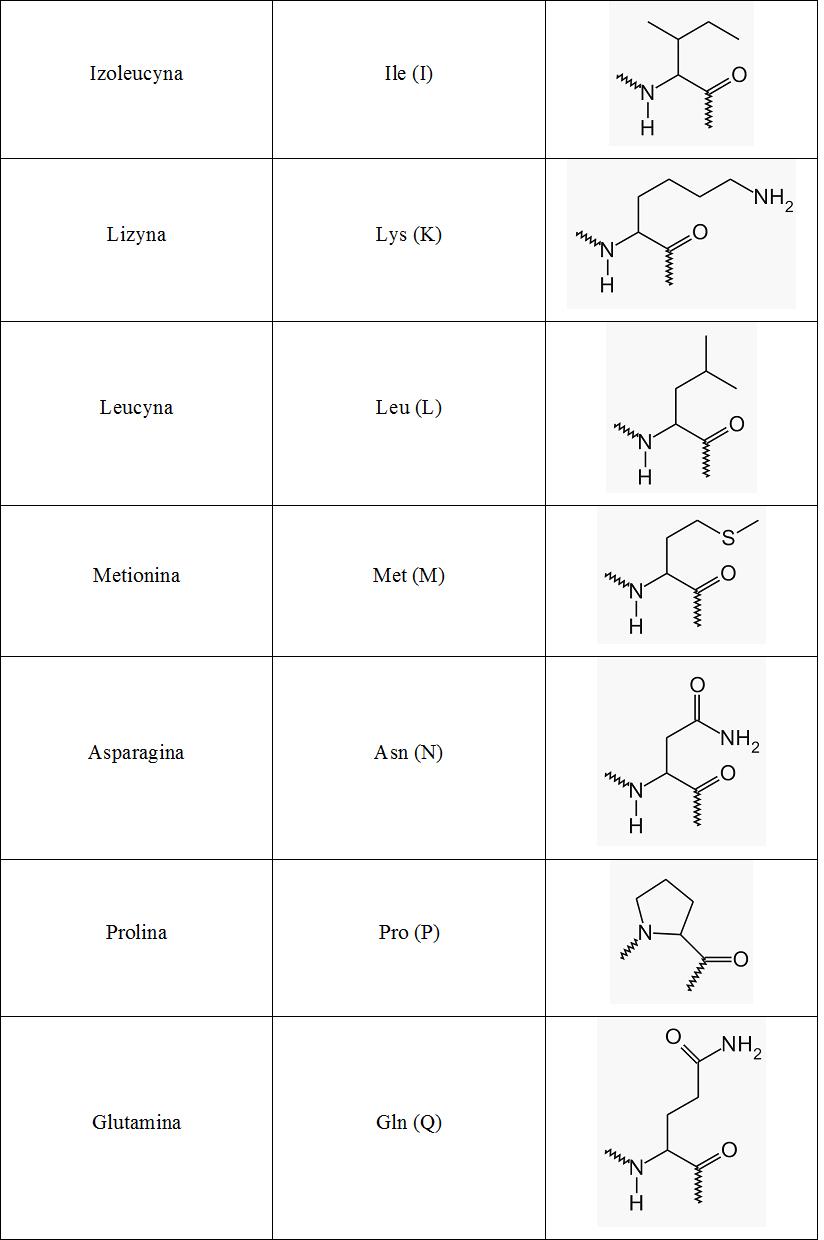
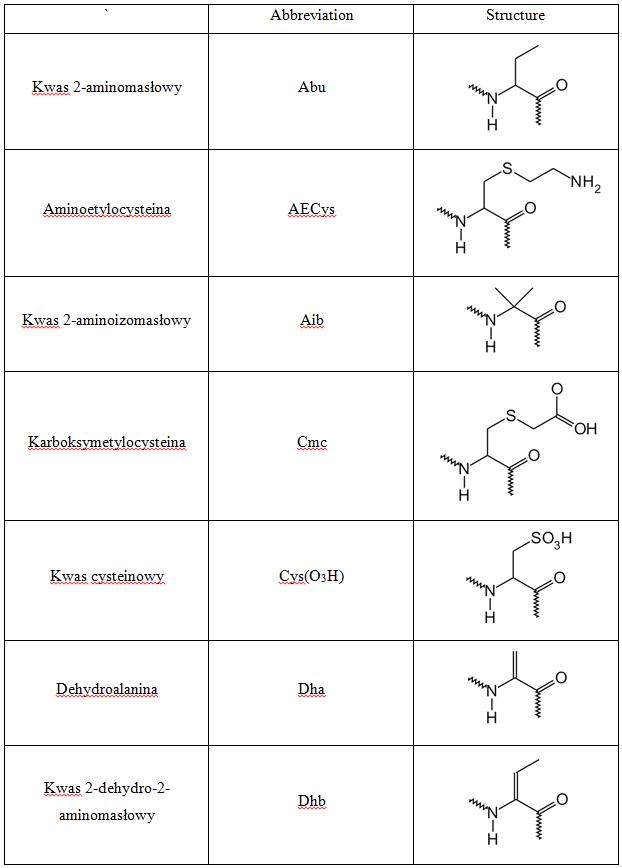
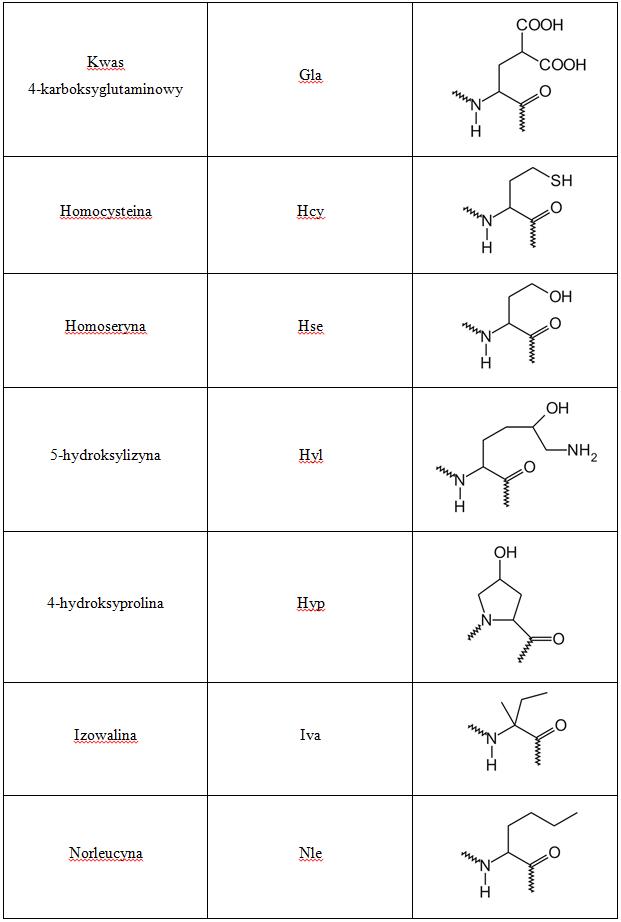
Białka składają się głównie z 20 tzw. białkowych reszt aminokwasowych. Ich łańcuchy boczne posiadają różne kształty, ładunek elektrostatyczny, aktywność chemiczną oraz wykazują zróżnicowane tendencje do tworzenia wiązań wodorowych. W białkach spotkać również można tzw. aminokwasy niebiałkowe, które są wprowadzane do sekwencji podczas procesów postranslacyjnych.
Z uwagi na swój charakter chemiczny aminokwasy mogą zostać podzielone na kilka grup. Jednym z takich podziałów, a zarazem najbardziej użytecznym, gdy mowa o strukturach i ich właściwościach, jest ten, w którym kryterium jest skład łańcucha bocznego aminokwasu. Opisany podział reszt aminokwasowych przedstawia się następująco:
- niepolarne, alifatyczne: Gly, Ala, Val, Leu, Ile i Pro,
- zawierające grupy hydroksylowe lub atomy siarki: Ser, Thr, Cys, Met,
- zawierające pierścienie aromatyczne: Phe, Trp, Tyr,
- zawierające grupy zasadowe: Lys, Arg, His,
- zawierające grupy kwasowe: Asp, Asn, Glu, Gln.
Ogólne informacje strukturalne
Sekwencja aminokwasowa oraz rozmieszczenie mostków disulfidowych w łańcuchu białkowym to tzw. struktura I rzędowa. Łańcuchy takie mogą tworzyć układy przestrzenne - helisy czy β-kartki. Są to elementy struktury II rzędowej. Wzajemne ich połączenie i umiejscowienie w przestrzeni to struktura III rzędowa. Z kolei oddziaływanie ze sobą kilku cząsteczek białka prowadzące do uzyskania dimerów, trimerów, tetramerów itd, to struktura IV rzędowa.
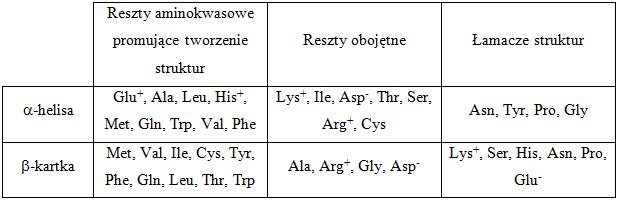
Według Chou i Fasmana, można wyróżnić aminokwasy promujące tworzenie α-helisy i β-kartki, neutralne oraz takie, które przeszkadzają w tworzeniu wymienionych struktur – tablica 3 [1].
Tablica 3. Tendencje reszt aminokwasowych do tworzenia i łamania struktur drugorzędowych
Przestrzenną strukturę zwiniętego białka stabilizują następujące czynniki: wiązania wodorowe, wiązania jonowe tzw. mostki solne, oddziaływania hydrofobowe, oddziaływania Van der Waalsa i mostki disulfidowe – schematycznie są one przedstawione na rysunku 2 [2].
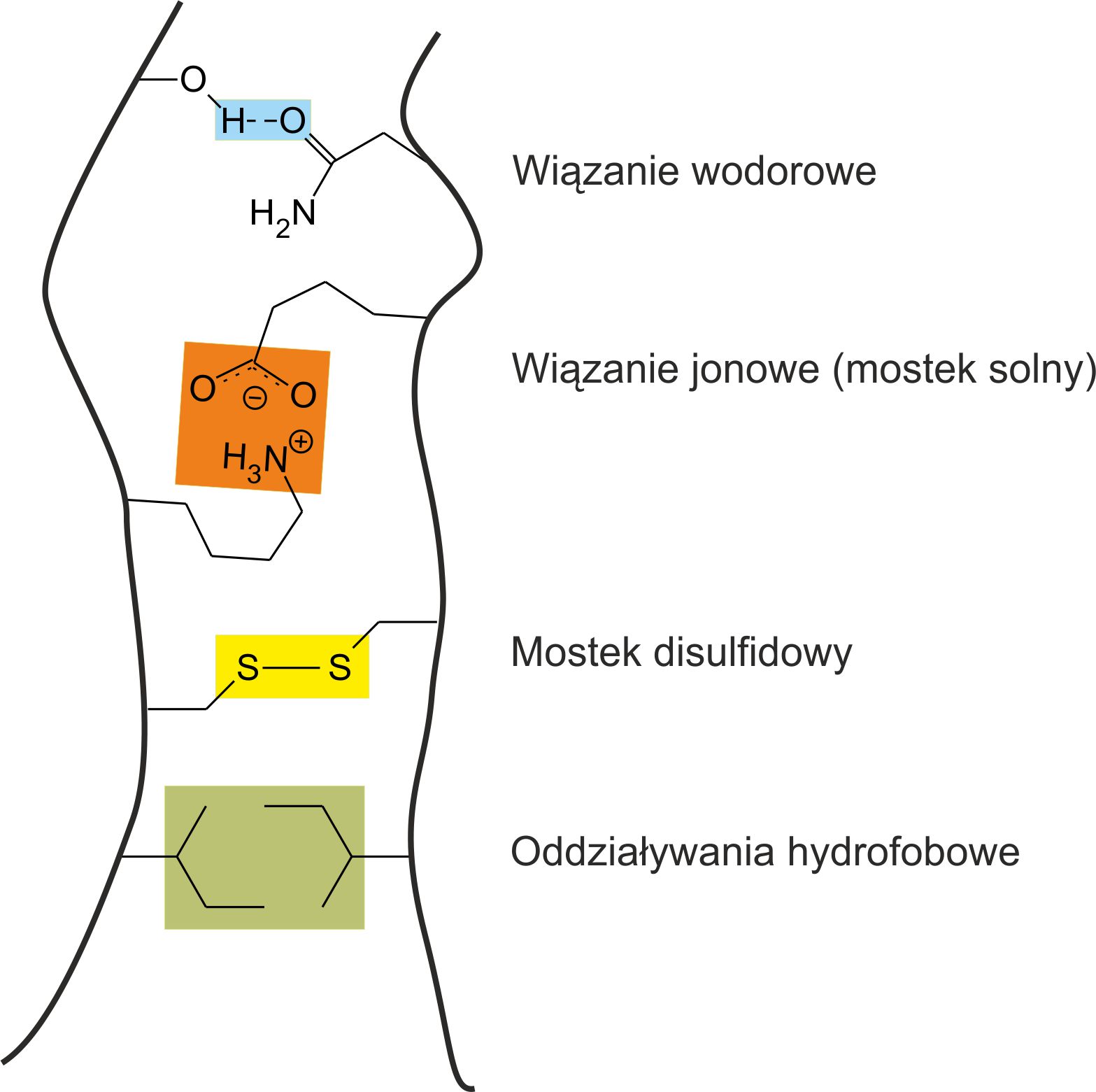
Rysunek 2. Oddziaływania stabilizujące struktury przestrzenne białek. Na podstawie [2]
Struktura drugorzędowa
Helisa
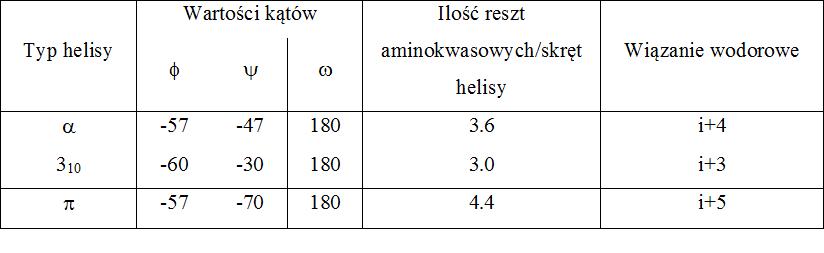
Helisa przypomina prawo lub lewoskrętną śrubę. Wyróżnić można kilka ich typów, z których każdy oznacza się zapisem N
n. Struktura helisy niezależnie od jej rodzaju stabilizowana jest wiązaniami wodorowymi. Dodatkowymi parametrami są: liczba reszt aminokwasowych przypadających na skręt N, ilość atomów łańcucha głównego przypadająca na pętlę spiętą wiązaniem wodorowym - n, skok helisy, czyli powtarzający się dystans (dla α-helisy to 0,54 nm) oraz odległość pomiędzy sąsiadującymi aminokwasami (0,15 nm dla α-helisy). Stosując powyższą nomenklaturę α-helisa to helisa 3,6
13. Oznacza to, że 3,6 reszt aminokwasowych przypada na skręt helisy, a pętlę tworzy 13 atomów łańcucha głównego. Charakterystyka różnych typów helis przedstawiona jest w tablicy 4 oraz na rysunku 3 [2].

Rysunek 3. Schemat wiązań wodorowych stabilizujących różne typu helis. Na podstawie [2]
Tablica 4. Charakterystyka różnych typów helis
β-kartka
Niektóre łańcuchy aminokwasowe nie są w stanie utworzyć wewnątrzłańcuchowych wiązań wodorowych, zamiast tego struktura białka stabilizowana jest międzyłańcuchowymi wiązaniami wodorowymi. Rezultatem tego jest rozciągnięta struktura przestrzenna zwana β-kartką. W porównaniu do helisy odległość pomiędzy sąsiadującymi aminokwasami w strukturze rozciągniętej wynosi 0,72 nm. Odpowiednie wartości kątów ϕ i ψ wynoszą -139° i 135° dla antyrównoległej β-kartki oraz -119° i 113° dla struktury równoległej. Oba przykłady pokazane są na rysunku 4 [3].

Rysunek 4. Reprezentacja dwóch typów β-kartki. Na podstawie [3]
Zgięcia
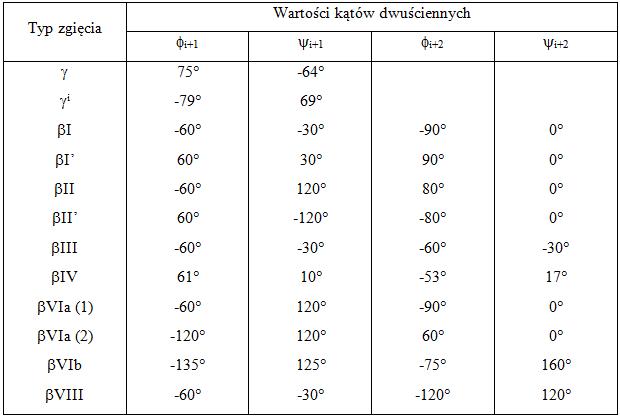
Wiele białek jest ciasno upakowanych w swojej strukturze przestrzennej i posiada globularny kształt. Jest to spowodowane występowaniem zgięć różnych typów: α, π, β i γ, w które jest zaangażowanych odpowiednio pięć, sześć, cztery i trzy reszty aminokwasowe. Najbardziej powszechnie występującymi są β i γ-zgięcia – rysunek 5 [2], tablica 5. Nazwa β-zgięcie pochodzi od antyrównoległej struktury β-kartki, gdzie dochodzi do zmiany kierunku łańcucha aminokwasowego.
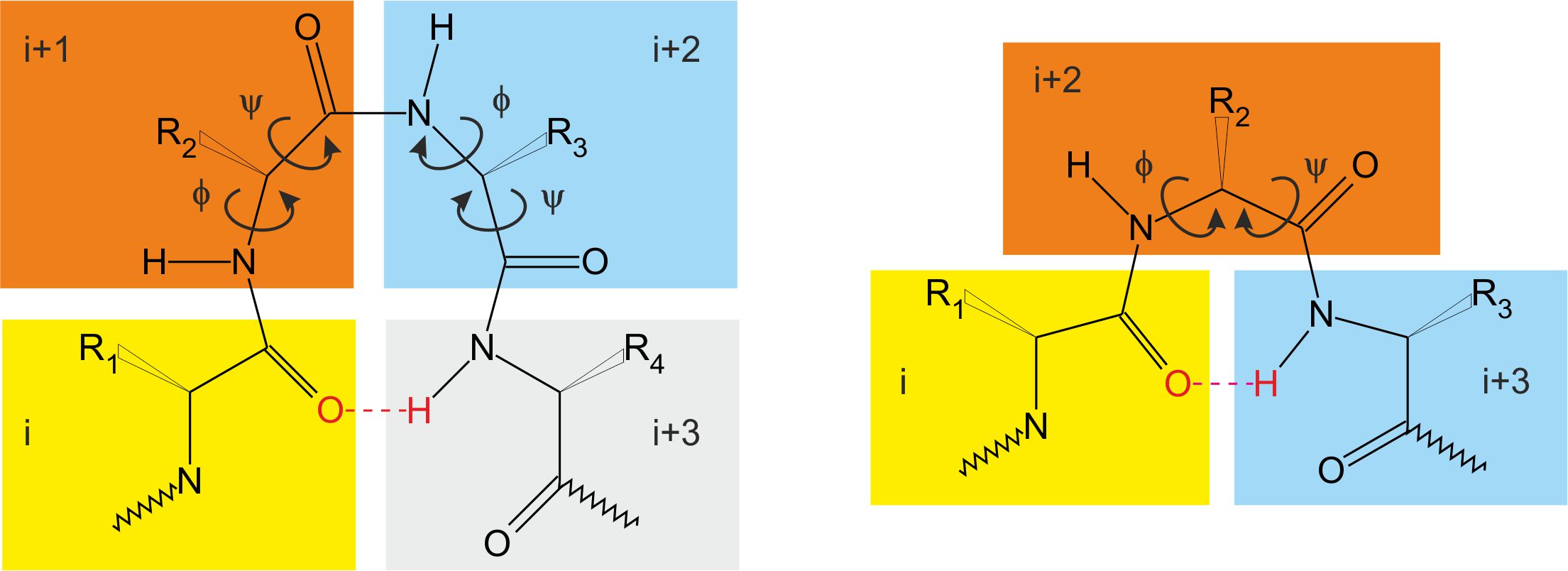
Rysunek 5. Schemat β (po lewej) oraz γ (po prawej) zgięcia. Na podstawie [2]
Tablica 5. Kąty torsyjne występujące w β i γ zgięciach [2]
Domeny strukturalne występujące w białkach
Analiza wielu struktur białek zaowocowała zdefiniowaniem tzw. struktur naddrugorzędowych zwanych inaczej domenami. Jest to wzajemne rozmieszczenie przestrzenne poszczególnych elementów struktury drugorzędowej względem siebie. Domeny strukturalne odpowiedzialne są również za właściwości biologiczne białek. Poniżej została przedstawiona krótka charakterystyka najczęściej występujących tych motywów strukturalnych.
Domena α (helisa)
To właściwie najbardziej powszechny motyw strukturalny. Białka, w których występują głównie struktury helikalne mogą mieć zarówno kształt globularny jak mioglobina – rysunek 6 [4], lub fibrylarny. Przykładem tego drugiego jest rodopsyna wołowa, będąca receptorem transbłonowym składającym się z siedmiu odcinków helikalnych – rysunek 7 [5]. Ich kluczowym elementem domenowym jest struktura tzw. zamka leucynowego – rysunek 8 [6].
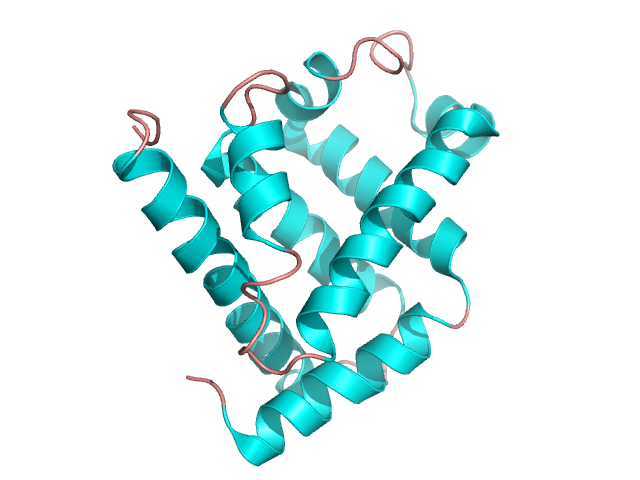
Rysunek 6. Struktura mioglobiny. Na podstawie struktury pdb [1mbn] [4]

Rysunek 7. Struktura rodopsyny wołowej. Na podstawie struktury pdb [1gzm] [5]
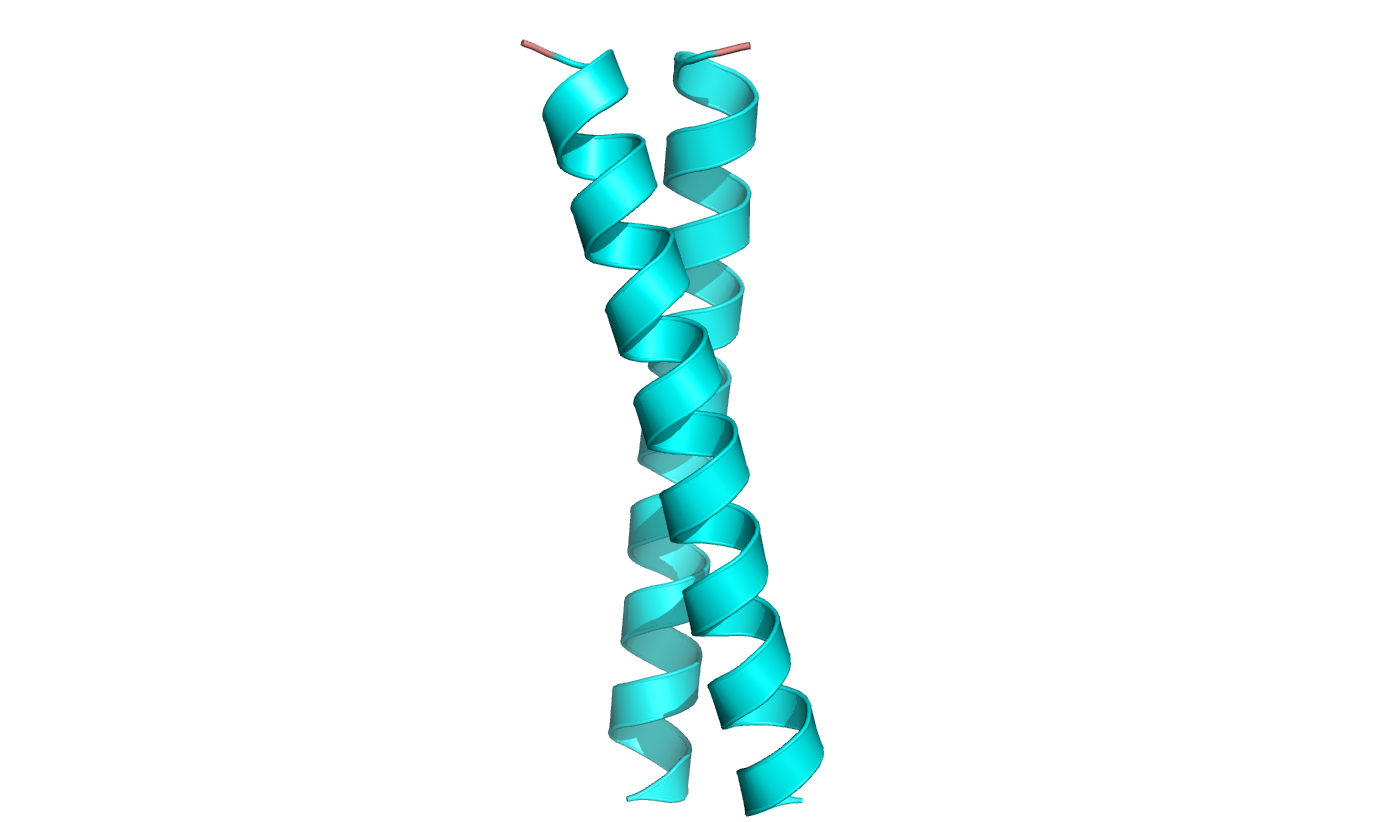
Rysunek 8. Zamek leucynowy, jeden z elementów receptorów transbłonowych. Na podstawie struktury pdb [1zij] [6]
Szczególnym przykładem domeny helikalnej jest helisa kolagenowa. To białko zbudowane jest z trzech helikalnych łańcuchów polipeptydowych po około 1000 reszt aminokwasowych każdy, skręconych wokół siebie – rysunek 9 [7].
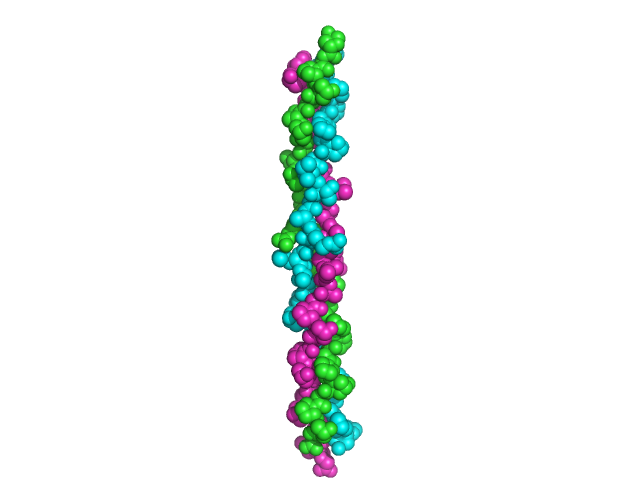 Rysunek 9. Helisa kolagenowa. Na podstawie struktury pdb [1bkv] [7]
Rysunek 9. Helisa kolagenowa. Na podstawie struktury pdb [1bkv] [7]Sekwencja aminokwasowa poszczególnych łańcuchów składa się z powtarzalnych fragmentów o sekwencji Gly-Pro-Hyp. Co ciekawe pojedyncze helisy nie są stabilizowane wewnątrzłańcuchowymi wiązaniami wodorowymi. To protony amidowe Gly tworzą je z akceptorem z drugiego łańcucha. Dodatkowo pierścienie pirolidynowe Pro i Hyp ułożone są w taki sposób, aby nie powodować zawad sterycznych.
Domena βαβ
To kombinacja dwóch struktur rozciągniętych przedzielonych helisą – rysunek 10.
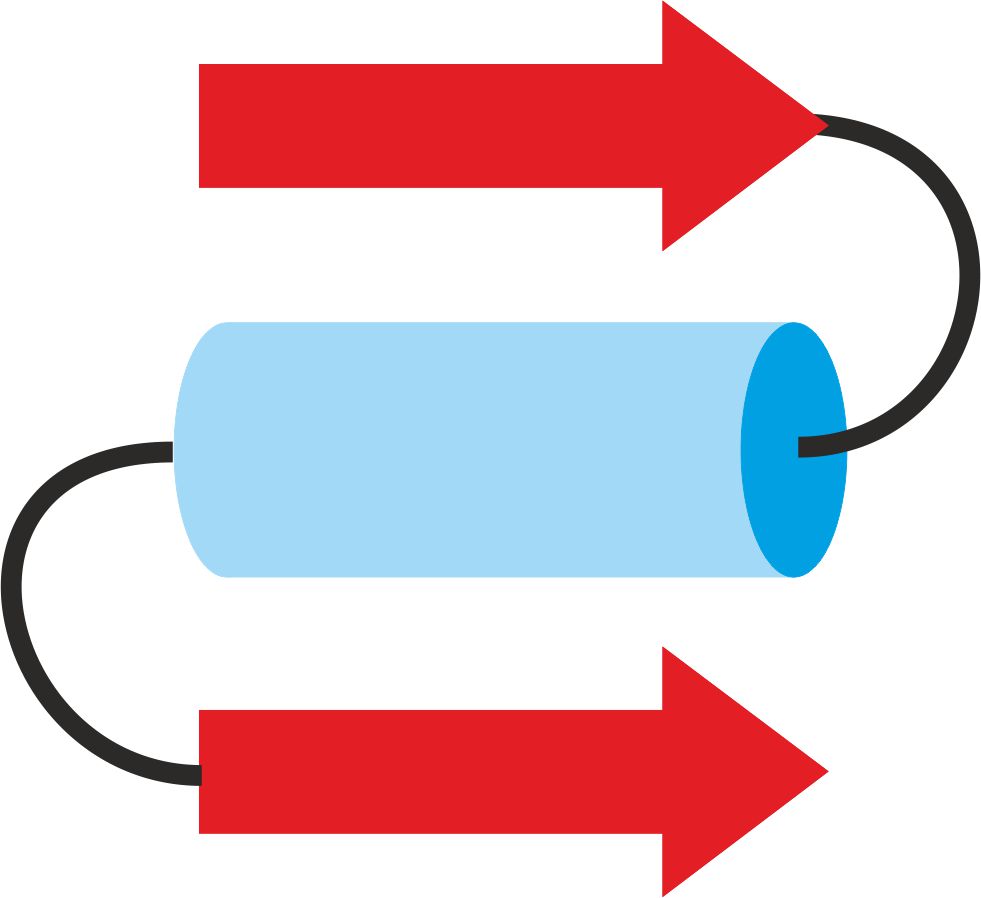
Rysunek 10. Schemat domeny βαβ
Domena taka występuje w jednej z dehydrogenaz – rysunek 11 [8], tworząc tzw. beczkę TIM.
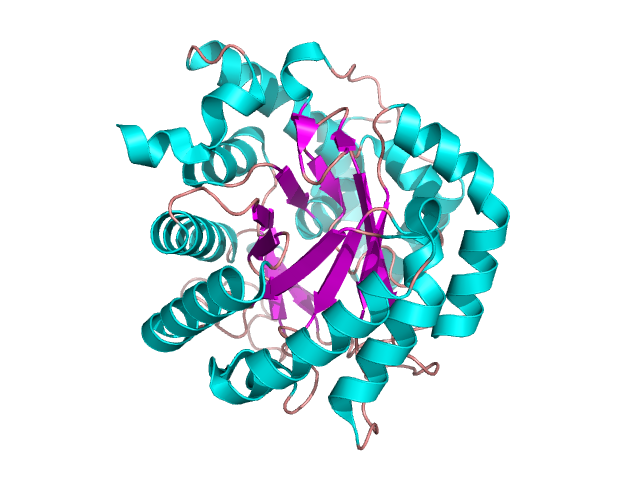
Rysunek 11. Struktura dehydrogenazy dihydroorotatowej. Na podstawie [2wv8] [8]
Domeny utworzone przez β-kartki
Obok domen helikalnych istnieje szereg złożonych struktur, które są utworzone przez rozciągnięte struktury β – rysunek 12.

Rysunek 12. Ułożenie łańcuchów β w różnych domenach: klucz grecki (po lewej), β-kanapka (środek), struktura antyrównoległa (po prawej)
Są to: klucz grecki – rys. 13 [9], kanapka β – rys. 14 [10], beczka β – rys. 15 [11], śmigło β – rys. 16 [12], siodło β – rys. 17 [13]. Wszystkie te motywy strukturalne charakteryzuje ciekawe ułożenie rozciągniętych struktur β. Pierwsza z wymienionych domen stabilizowana jest połączeniem dwóch skrajnych β-kartek w jednostce składającej się z czterech rozciągniętych łańcuchów. Kanapka β z kolei to wielopoziomowa konstrukcja, w której łańcuchy z jednego poziomu połączone są z niższym, co gwarantuje stabilność utworzonej domeny. Pozostałe wymienione domeny β to antyrównoległa β-kartka. W zależności od właściwości białka przybiera ona struktury pokazane na rysunkach.
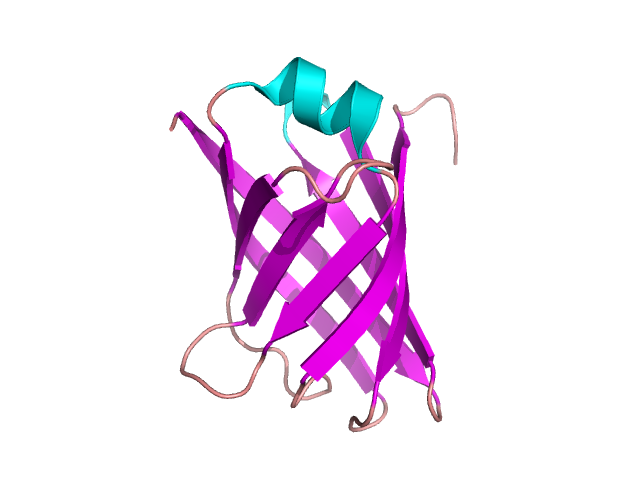
Rysunek 13. Struktura białka VAPB – zawierającego motyw klucza greckiego. Na podstawie struktury pdb [4cv7] [9]
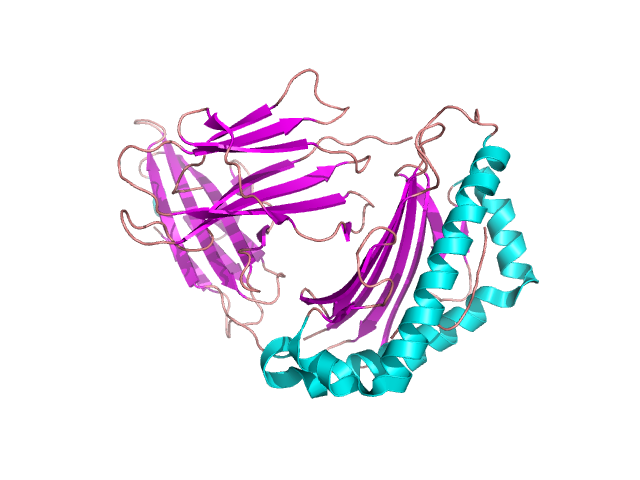
Rysunek 14. Struktura β-2-globuliny, w której znajduje się domena β-kanapki. Na podstawie struktury pdb [3bgm] [10]
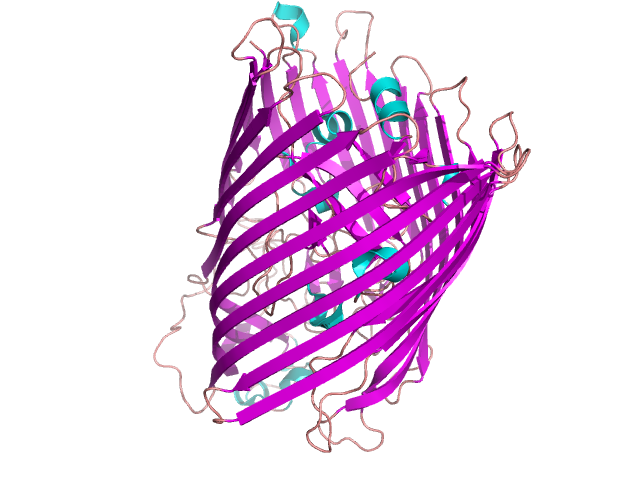
Rysunek 15. Domena β-beczki znajdującej się w białku FHUA. Na podstawie [1fi1] [11]
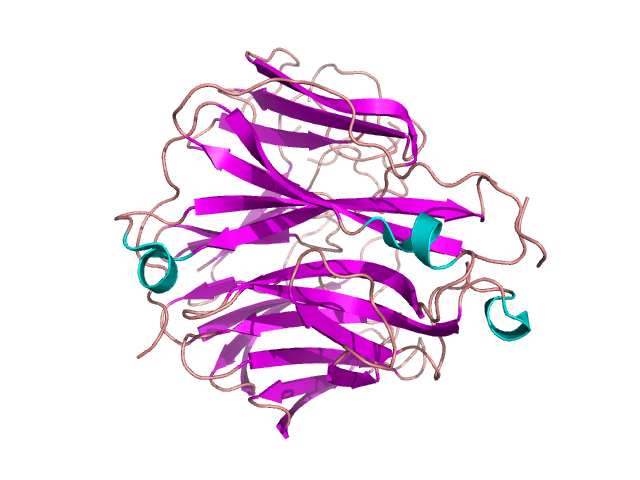
Rysunek 16. Struktura neuramidazy z domeną śmigła w roli głównej. Na podstawie [3cl0] [12]
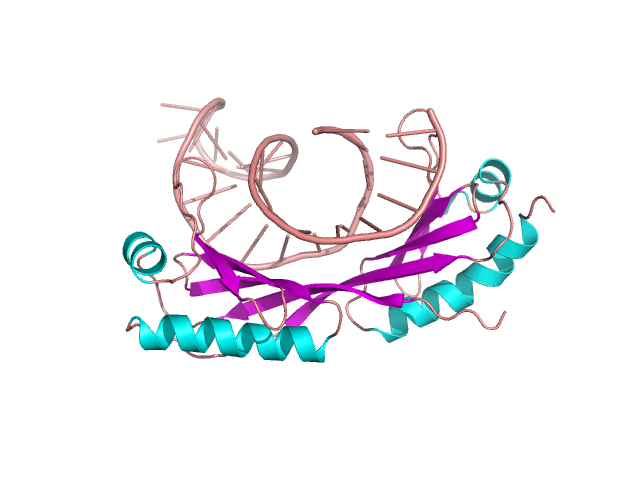
Rysunek 17. Kompleks DNA z białkiem TATA. Na podstawie struktury pdb [1ytb] [13]
Palec cynkowy
Ten element strukturalny obecny jest w białkach wiążących DNA. Domena ta składa się z 30 aminokwasów tworzących dwa łańcuchy antyrównoległej β-kartki i jedną α-helisę. Obecność jonu Zn
2+ jest niezbędna do stabilizacji całej struktury. Palec cynkowy to ewolucyjnie zakonserwowany element strukturalny białek. Fragment helikalny znajduje się na C-końcu, z kolei łańcuch β tworzy odcinek N-końcowy. Kation cynku jest helatowany przez dwie reszty histydyny, które są częścią fragmentu helikalnego i dwie reszty cysteiny znajdujące się w β-kartce – rysunek 18 [14].
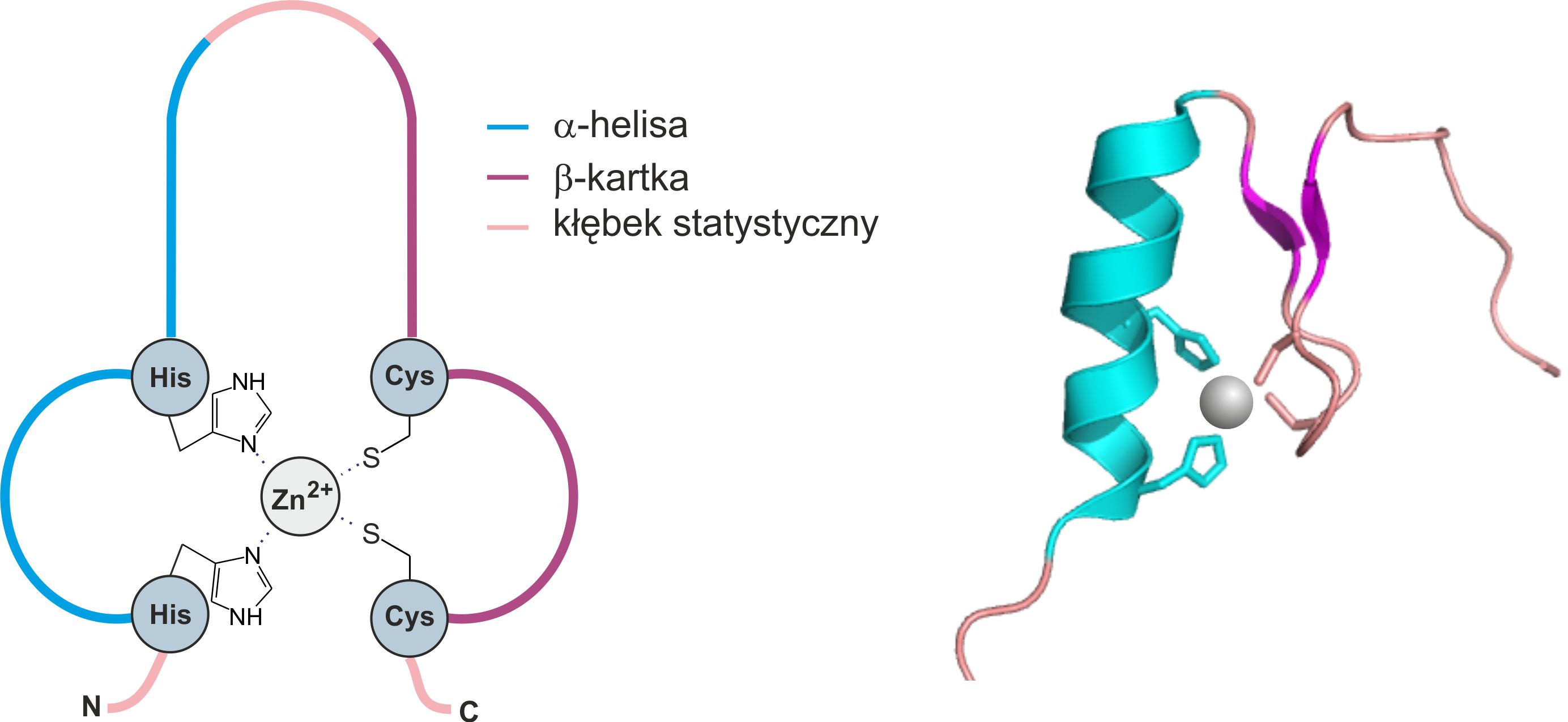
Rysunek 18. Domena palec cynkowy. Na podstawie [1x5w] [14]
Złożoność konformacyjna białek nie jest tematem zamkniętym. Przeglądając bazę struktur tych biopolimerów, uwagę zwraca ich niewiarygodna ilość. Wszystkie one zbudowane są z przedstawionych powyżej elementów struktury drugorzędowej lub kombinacji opisanych domen strukturalnych. Mnogość ta świadczy o wielkiej różnorodności i wysokim poziomie skomplikowania materii. Z drugiej zaś strony jest to gwarancja dla chemików strukturalnych, że przez długi czas będą znajdować obiekty swoich badań.
Literatura:
[1] G.C. Barret, D.T. Elmore. Amino Acids and Peptides, Cambridge University Press, UK (2004).
[2] N. Sewald, H.D. Jakubke. Peptides: Chemistry and Biology, Wiley-VCH Verlag GmbH (2002).
[3] J.M. Berg, J.L. Tymoczko, L. Stryer. Biochemistry, Fifth Edition, W.H. Freeman, New York (2002).
[4] H.C. Watson, The Stereochemistry of the Protein Myoglobin, Prog. Stereochem., 4, 299 (1969).
[5] J. Li, P. Edwards, M. Burghammer, C. Villa, G.F.X. Schertler, Structure of Bovine Rhodopsin in a Trigonal Crystal Form, J. Mol. Biol., 343, 1409 (2004).
[6] E.K. O’Shea, J.D. Klemm, P.S.Kim, T. Alber, X-Ray Structure of the GCN4 Leucine Zipper, a Two Stranded, Parallel Coiled Coil, Science, 254, 539 (1991).
[7] R.Z. Kramer, J. Bella, P. Mayville, B. Brodsky, H.M. Berman, Sequence Depended Conformational Variations of Collagen Triple-Helical Structure, Nat. Struct. Biol., 6, 454 (1999).
[8] I. Fritzson, B. Svenson, S.Al-Karadaghi, B. Walse, U. Wellmar, U.J. Nillsson, D. Da GracaThrige, S. Johnson, Inhibition of Human Dhoby by 4-Hydroxycoumarins, Fenamic 2 Acids, and N-(Alkylcarbonyl)anthranilic Acid Identified by Structure-Guided Fragment Selection, Chemmedchem, 5, 608 (2010).
[9] C. Geerds, J. Wohlmann, A. Haas, H.H. Niemann, Structure of Rhodococcus Equi Virulence-Associated Protein B (VAPB) Reveals an Eight-Stranded Antiparallel [Beta]-Barrel Consisting of Two Greek-Key Motifs, Acta Crystallogr., Sect. F, 70, 866 (2014).
[10] F. Mohammed, M. Cobbold, A.L. Zarling, M. Salim, G.A. BarrettiWilt, J. Shabanowitz, D.F. Hunt, V.H. Engelhard, B.E. Willcox, Phosphorylation-Dependent Interaction Between Antigenic Peptides and MHC Class I: A Molecular Basis for the Presentation of Transformed Self, Nat. Immunol., 9, 1236 (2008).
[11] A.D. Ferguson, J. Kodding, G. Walker, C.Bos, J.W. Coulton, K. Diederichs, V. Braun, W. Welte, Active Transport of an Antibiotic Rifsmycin Derivative by the Outer-Membrane Protein FHUA, Structure, 9, 707 (2001).
[12] P.J. Collins, L.F. Haire, Y.P. Lin, J. Liu, R.J. Russell, P.A. Walker, J.J. Skehel, S.R., Martin, A.J. Hay, S.J. Gamblin, Crystal Structures of Oseltamivir-Resistant Influenza Virus Neuramidase Mutants, Nature, 453, 1258 (2008).
[13] Y. Kim, J.H. Geiger, S. Hahn, P.B. Sigler, Crystal Strycture of a Yeat TBP/TATA-Box Complex, Nature, 512, (1993).
[14] M. Yoneyama, S. Koshiba, N. Tochio, M. Inoue, T. Kigawa, S. Yokoyama, Solution Structure of the C2H2Type Zinc-Binding Domain of Human Zinc Finger Protein 64, Isoforms 1 and 2 w druku.
 Z uwagi na szeroki zakres właściwości biologicznych białek, kluczowe jest zrozumienie zależności aktywność-struktura. Poznanie budowy przestrzennej całej cząsteczki bądź jej fragmentów odpowiedzialnych za aktywność to wyzwanie dzisiejszej biochemii strukturalnej. Znajomość szczegółów strukturalnych pozwala na zrozumienie mechanizmu działania oraz dynamicznego zachowania dużych cząsteczek pod wpływem różnych warunków.
Z uwagi na szeroki zakres właściwości biologicznych białek, kluczowe jest zrozumienie zależności aktywność-struktura. Poznanie budowy przestrzennej całej cząsteczki bądź jej fragmentów odpowiedzialnych za aktywność to wyzwanie dzisiejszej biochemii strukturalnej. Znajomość szczegółów strukturalnych pozwala na zrozumienie mechanizmu działania oraz dynamicznego zachowania dużych cząsteczek pod wpływem różnych warunków.