Marta Cyman
Gdański Uniwersytet Medyczny
strony wersji drukowanej: 22-27

strony wersji drukowanej: 22-27

Metoda qPCR - wprowadzenie
Ilościowa łańcuchowa reakcja polimerazy (ang. quantitative polimerase chain reaction, qPCR), zwana również jako PCR w czasie rzeczywistym (ang. Real-time PCR), jest wariantem klasycznej reakcji PCR opracowanej w 1983 roku przez Mullisa i wsp. Podstawowa reakcja PCR pozwala na syntezę bardzo dużych ilości kopii DNA genomowego. Istotą tego procesu jest powielanie określonej, poszukiwanej sekwencji za pomocą enzymu polimerazy DNA i starterów komplementarnych do interesującego nas fragmentu matrycowego DNA. Ponadto w mieszaninie reakcyjnej powinny zostać zawarte substraty do wydłużania nici DNA (trifosforany deoksyrybonukleotydów, dNTPs) oraz jony Mg2+, niezbędne dla prawidłowego funkcjonowania polimerazy DNA. Reakcję przeprowadza się w termocyklerze, który pozwala na szybkie zmiany temperatur. Pierwszym etapem reakcji jest podgrzanie mieszaniny reakcyjnej do temperatury ok. 95°C w celu denaturacji (rozpadu) podwójnej nici DNA. Następnie dochodzi do obniżenia temperatury do wartości charakterystycznej dla danej pary starterów (zazwyczaj w przedziale 45°C - 70°C). Na tym etapie startery przyłączają się do matrycy. Po podniesieniu temperatury do ok. 72°C do matrycy ze starterami przyłącza się polimeraza DNA i rozpoczyna syntezę nowych, komplementarnych nici DNA. Cykl powtarzany jest wielokrotnie (zazwyczaj ok. 30-40 razy) do momentu uzyskania oczekiwanej ilości kopii powielanego fragmentu DNA. Detekcji dokonuje się poprzez rozdział elektroforetyczny produktu lub hybrydyzację. Klasyczny PCR, pomimo swoich zalet, nie pozwala na uzyskanie precyzyjnych wyników ilościowych.
Metody detekcji przyrostu produktu w reakcji qPCR
W pierwszej połowie lat dziewięćdziesiątych Higuchi i wsp. opracowali wariant pozwalający na monitorowanie przyrostu produktu w każdym cyklu reakcji. Zostało to osiągnięte dzięki zastosowaniu znacznika – bromku etydyny interkalującego do dwuniciowej struktury DNA. Termocykler wyposażony w źródło promieniowania UV i kamerę rejestrującą fluorescencję umożliwił pomiar wzrostu natężenia fluorescencji odpowiadającemu przyrostowi produktu. Pozwala to na oszacowanie stężenia amplikonu w trakcie reakcji. Kolejnym etapem badań nad techniką qPCR było wprowadzenie mniej toksycznych znaczników. Obecnie najczęściej wykorzystywany jest SYBR Green I lub wyznakowane fluorochromami sondy molekularne komplementarne do syntetyzowanych sekwencji.
Metody niespecyficzne
W porównaniu do bromku etydyny barwnik fluorescencyjny SYBR Green I jest bardziej czuły i specyficzny, ponieważ przyłącza się tylko do dwuniciowej struktury DNA (dsDNA), nie wiąże się z DNA jednoniciowym (ssDNA). Wynikiem połączenia barwnika z dsDNA jest wzrost fluorescencji przy długości światła 520 nm po wzbudzeniu wiązką od długości fali w przedziale od 480 do 494 nm.
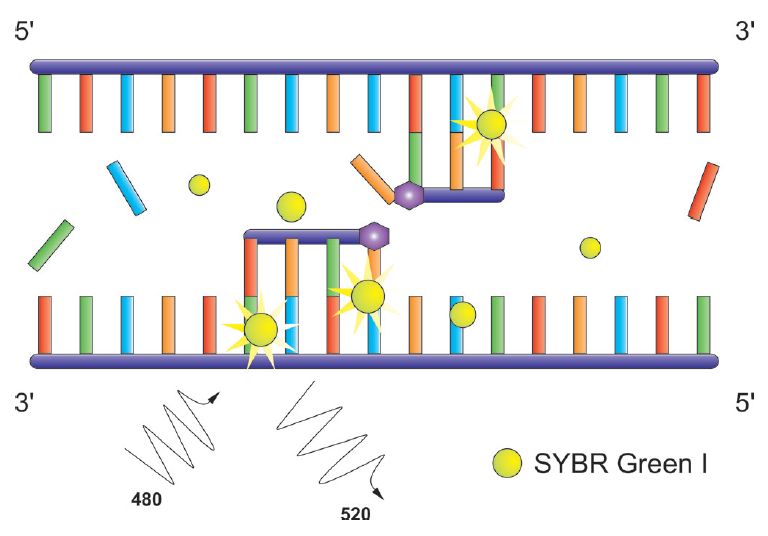
Ryc. 1. Zasada działania barwnika fluorescencyjnego SYBRGreen I.
Wadą SYBR Green I jest wiązanie się do każdej dwuniciowej struktury, również do sekwencji niespecyficznych np. połączeń starterów (primer-dimer). W przypadku stosowania tego barwnika należy po reakcji przeprowadzić analizę krzywej topnienia w celu wyeliminowania nieprawidłowości.
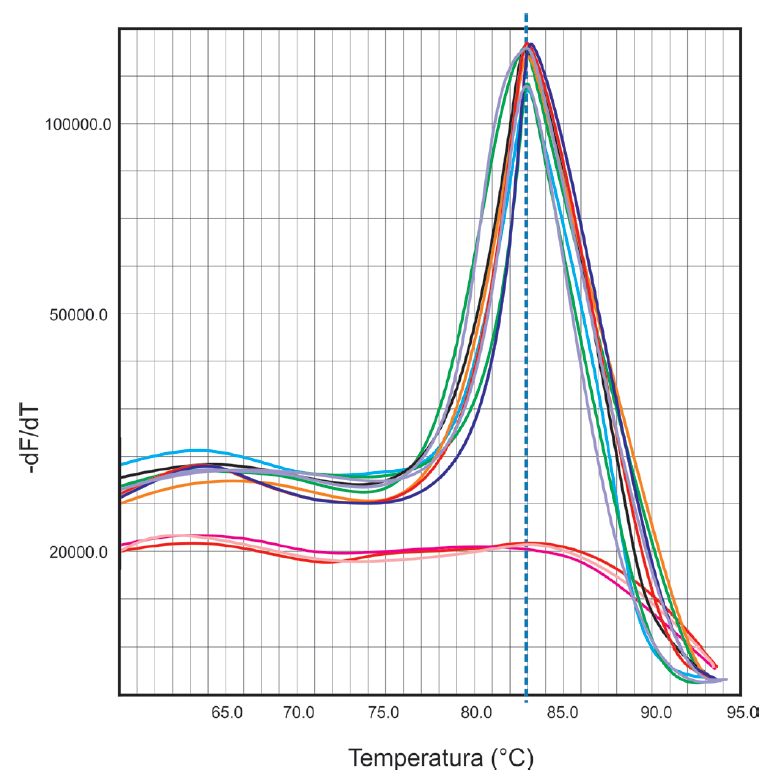
Ryc. 2. Krzywe topnienia dla produktów reakcji qPCR. Oś OY – ujemna zmiana fluorescencji, oś OX – zmiany temperatury.
Wadą SYBR Green I jest wiązanie się do każdej dwuniciowej struktury, również do sekwencji niespecyficznych np. połączeń starterów (primer-dimer). W przypadku stosowania tego barwnika należy po reakcji przeprowadzić analizę krzywej topnienia w celu wyeliminowania nieprawidłowości.
Metody specyficzne
Aby wyeliminować problem specyficzności fluorescencji syntetyzowanego produktu do reakcji qPCR stosowane są sondy molekularne znakowane fluorochromem m.in. Fluorescence Resonance Energy Transfer (FRET), TaqMan, Molecular Beacons. Wśród tych metod najszerzej wykorzystuje się sondy TaqMan. Zasada działania sondy TaqMan opiera się na hydrolizie pojedynczej sondy znakowanej podwójnie. Na końcu 5‘ sondy znajduje się fluorochrom, zwany reporterem (np. 6-karboksyfluoresceina), natomiast na końcu 3‘ znajduje się wygaszacz (np. 6-karboksytetrametylrodamina).
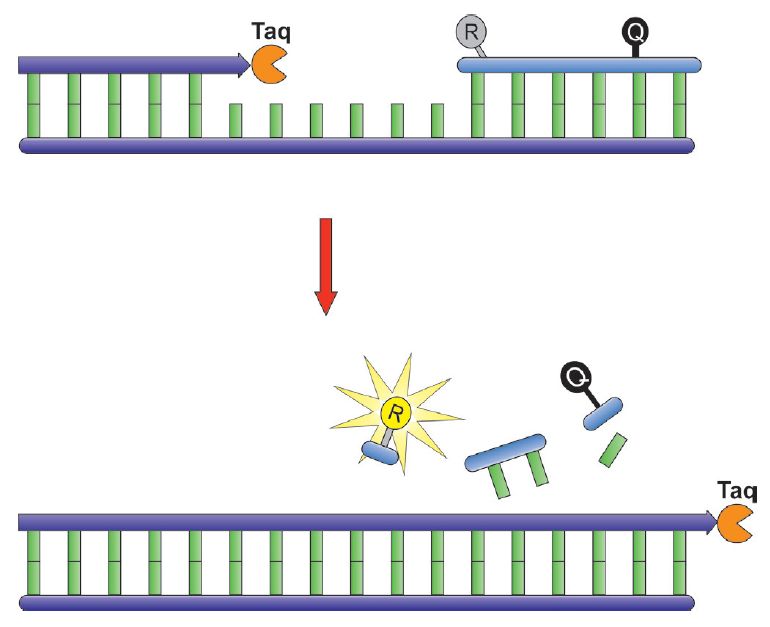

Ryc. 3. Zasada działania sondy TaqMan (Taq – polimeraza Taq, R – reporter, Q – wygaszacz).
Dopóki oba związki są połączone z liniową sondą i znajdują się blisko siebie fluorescencja jest tłumiona. W momencie gdy w trakcie elongacji dojdzie do degradacji sondy przez polimerazę Taq, posiadającą aktywność 5‘-egzonukleazy, uwolniony fluorochrom emituje flurescencję. Do hydrolizy sondy TaqMan i tym samym do emisji fluorescencji może dojść jedynie podczas elongacji wybranego fragmentu DNA. Można założyć, że fluorescencja pochodzi tylko i wyłącznie od oczekiwanego amplikonu.
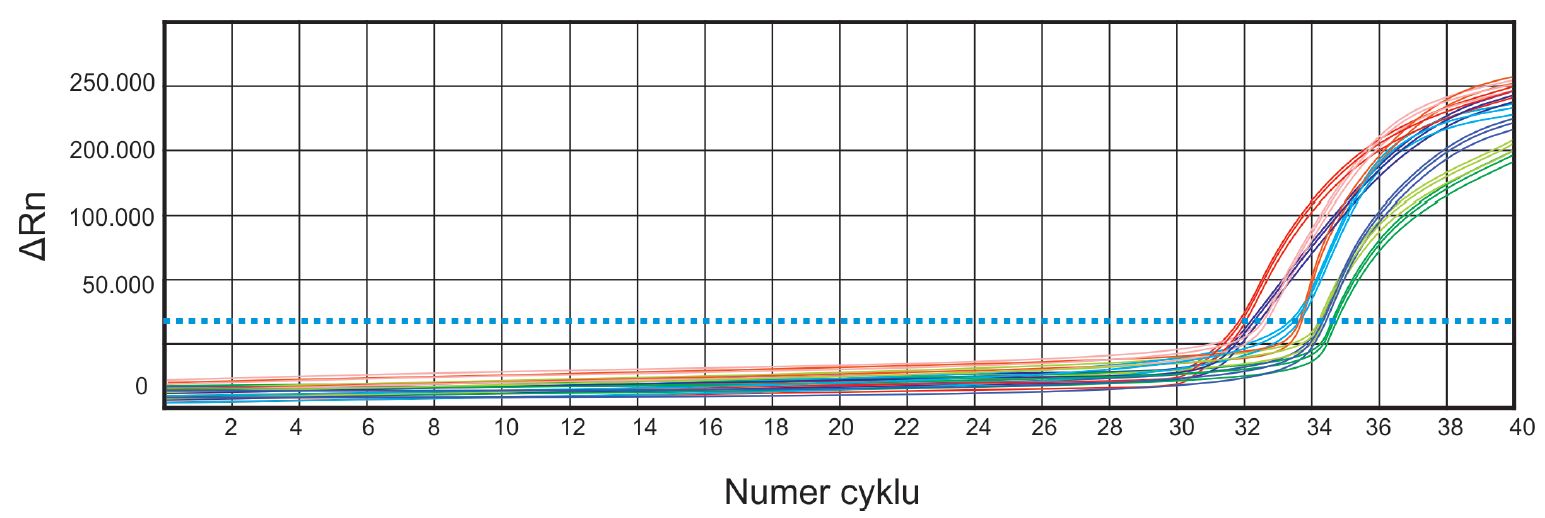
Ryc. 4. Wykres zależności nasilenia sygnału fluorescencji SYBRGreen I (oś OY) od numeru cyklu (oś OX). Niebieską, przerywaną linią zaznaczono moment, w którym reakcja qPCR wkracza w fazę logarytmicznego wzrostu.
Matryca
Oprócz starannie zaprojektowanych starterów i metody detekcji produktu należy również zadbać o jakość matrycy. Matrycą do reakcji qPCR może być DNA oraz oraz RNA gdy chcemy uzyskać informację np. o ekspresji danego genu. Jednak, aby móc włączyć do reakcji materiał genetyczny w postaci RNA należy najpierw przepisać informację na nim zawartą na DNA komplementarne (cDNA). Wykorzystanie cDNA jako matrycy pozwala na powielanie tylko sekwencji kodujących, z pominięciem intronów. Syntezę cDNA z matrycy RNA prowadzi się w obecności enzymu odwrotnej transkryptazy oraz polimerazy DNA. Dzięki zastosowaniu odpowiedniego zestawu starterów możliwe jest uzyskanie matrycy w postaci cDNA zsyntetyzowanej tylko z cząsteczki mRNA, z pominięciem rRNA i tRNA. Taka modyfikacja metody qPCR nosi nazwę RT-PCR (ang. Reverse transcriptase PCR).
Metody analizy wyników uzyskanych podczas reakcji qPCR
Istotą reakcji qPCR jest założenie, że ilość produktu wzrasta wykładniczo wraz z każdym cyklem reakcji. W praktyce, ze względu na stopniowe wyczerpanie się substratów do reakcji, spadek aktywności polimerazy czy też konkurencję o matrycę pomiędzy starterami i produktem, wydajność reakcji z czasem się obniża. Z tego powodu każda reakcja PCR osiąga w pewnym momencie fazę plateau, w której produktu już nie przybywa. Właściwa analiza przyrostu produktu opiera się na określeniu tzw. cyklu progowego (Ct ang. threshold cycle), czyli cyklu w którym fluorescencja przekracza poziom tła. Numer cyklu, który definiuje Ct jest najczęściej odzwierciedleniem wejścia reakcji w fazę logarytmicznego przyrostu ilości amplikonu. Im niższy jest numer cyklu Ct tym większe stężenie stężenie początkowe matrycy wziętej do reakcji. Istnieją dwie metody analizy uzyskanych wyników. Pierwsza z metod opiera się na analizie krzywej standardowej, uzyskanej dla próbek o znanym stężeniu. Następnie aparat wylicza na jej podstawie stężenie DNA w matrycy. Drugim sposobem analizy danych jest względne oznaczenie ilościowe przez porównanie wartości Ct dla próbek badanych i genu referencyjnego (ΔCt). Następnie uzyskaną wartość ΔCt dla grupy badanej porównuje się z grupą kontrolną (ΔΔCt).
Znaczenie doboru właściwego genu referencyjnego
Z założenia gen referencyjny cechuje się stałą ekspresją bez względu na czynniki biologiczne. Najczęściej stosowanymi genami są: dehydrogenaza gliceraldehydo-3-fosforanowa (GAPDH), β-aktyna, cyklofilina, fosforybozylotransferaza hipoksantynowo-guaninowa (HPRT). Istotne jest przeprowadzenie badań mających na celu wyłonienie najbardziej stabilnego genu referencyjnego dla danej grupy badanej. Dobór przypadkowego genu konstytutywnego może skutkować błędnymi wynikami analizy qPCR. Obecnie uważa się, że nie istnieje jeden idealny gen kontrolny spełniający wszystkie kryteria uniwersalnej kontroli wewnętrznej. Ekspresja każdego z nich ulega w jakimś stopniu regulacji. Aby gen mógł być uznany za uniwersalny gen referencyjny powinien utrzymywać stałą ekspresję we wszystkich typach komórek i we wszystkich warunkach. Biorąc pod uwagę znaczenie właściwego doboru genu kontrolnego we wstępnym etapie projektowania doświadczenia należy porównać stabilność genów-kandydatów. Uważa się, że stosowanie kombinacji pary genów referencyjnych jest najkorzystniejsze podczas badań prowadzonych przy pomocy metody qPCR.
Zalety i wady qPCR
Niewątpliwą zaletą metody qPCR w porównaniu do klasycznej reakcji PCR jest możliwość pomiaru stężenia produktu w trakcie trwania reakcji bez potrzeby pracochłonnego etapu odczytu danych jakim jest rozdział elektroforetyczny czy hybrydyzacja. Dzięki temu, że pomiar i odczyt wyniku wyniku zachodzi w termocyklerze istnieje niskie ryzyko kontaminacji próbki. Krótszy czas reakcji oraz mniej skomplikowana procedura w porównaniu do podstawowej reakcji PCR wyjaśnia popularność w stosowaniu tej metody w badaniach nad DNA i RNA. Pomimo niepodważalnych zalet metoda qPCR posiada pewne ograniczenia. Koszt aparatury jest wysoki dlatego wciąż nie jest to metoda z wyboru w diagnostyce rutynowej. Ponadto parametrami krytycznymi są: etap projektowania eksperymentu, zaprojektowanie i dobór odpowiednich starterów, wybór odpowiedniego genu referencyjnego oraz przygotowanie materiału i analiza danych. Ze względu na wysoką czułość wykrywania nawet niewielkich ilości kwasów nukleinowych qPCR znalazło zastosowanie głównie w diagnostyce zakażeń wirusowych i bakteryjnych.
Zastosowanie qPCR w diagnostyce wirusologicznej
W wirusologii wykorzystuje się qPCR do pomiaru liczby kopii wirusa. Jest to kluczowy parametr pozwalający na ocenę skuteczności leczenia, ponieważ zaawansowanie choroby zależy od stopnia wiremii. Metoda ta może również służyć do diagnostyki zakażenia w początkowej fazie infekcji gdy przeciwciała jeszcze nie są wykrywalne. Monitorowanie wiremii w czasie jest szczególnie ważne w przypadku długotrwałego leczenia przeciwwirusowego w zakażeniach wywołanych m.in. HCV czy HIV. Wirusami oznaczanymi przy pomocy Real-time PCR są: wirus zapalenia wątroby typu B (WZW B, ang. hepatitis B virus, HBV), wirus zapalenia wątroby typu C (WZW C, ang. hepatitis C virus, HCV), wirus cytomegalii (ang. cytomegalovirus, CMV), ludzki wirus niedoboru odporności (ang. human immunodeficiency virus, HIV), wirus Epsteina-Barr (EBV). W przypadku pacjentów poddanych immunosupresji monitoruje się obecność materiału genetycznego pochodzącego od enterowirusów czy parwowirusa B19.
Zastosowanie qPCR w diagnostyce parazytologicznej
Real-time PCR w diagnostyce parazytologicznej służy do precyzyjnego określania stopnia inwazji przez pasożyty np. Toxoplasma gondii nie tylko we krwi, ale także w płynie owodniowym czy płynie mózgowo-rdzeniowym. Technikę tę można również wykorzystać w przypadku zakażenia pierwotniakami Entamoeba histolityca czy Plasmodium sp.
Zastosowanie qPCR w diagnostyce bakteriologicznej
Również w przypadku diagnostyki zakażeń bakteryjnych swoje zastosowanie znalazła metoda qPCR. Nie zastąpi ona tradycyjnej hodowli bakteryjnej jednak może służyć do szybkiego potwierdzenia lub wykluczenia infekcji. Pozwala to na niezwłoczne wdrożenie celowanego leczenia antybiotykami. Szczególnie przydatne jest wykorzystanie techniki qPCR w celu identyfikcji drobnoustrojów takich jak Mycobacterium tuberculosis, które w klasycznej hodowli namnażają się wolno lub wcale. Ponadto Real-time PCR może być stosowany do diagnostyki infekcji wywołanych przez: Legionella pneumophilia, Neisseria gonorrhoeae, Bacillus anthracis, Campylobacter sp., Chlamydia trachomatis, Listeria monocytogenes, Mycobaterium paratuberculosis czy Salmonella sp. Możliwa jest również analiza mutacji, która pozwala na monitorowanie oporności na antybiotyki takich drobnoustrojów jak: Staphylococcus aureus, Staphylococcus epidermidis, Helicobacter pylori, Enterococcus feacalis, Enterococcus feacium. Należy jednak pamiętać o ogarniczeniu metody wynikającym z tego, że qPCR wykrywa sekwencje DNA zarówno z żywych jak i martwych patogenów.
Zastosowanie qPCR w diagnostyce onkologicznej
PCR w czasie rzeczywistym jest wykorzystywany w diagnostyce i monitorowaniu chorób nowotworowych. Szczególnie istotną rolę owa technika odgrywa w nowotworach układu krwiotwórczego. Real-time PCR jest rutynowo stosowany w monitorowaniu poziomu ekspresji transkryptu BCR-ABL w przewleklej białaczce szpikowej, PML-RARA w ostrej białaczce szpikowej czy przy określaniu miana mutacji JAK2 w przewlekłych nowotworach mieloproliferacyjnych. Jest również podstawą do monitorowania minimalnej choroby resztkowej (MRD ang. minimal residual disease). W przypadku nowotworów złośliwych qPCR stosowany jest do oceny ilości kopii genów np. HER2 w nowotworach piersi oraz w przypadku guzów litych – do oceny ekspresji genów np. K-RAS w raku jelita grubego. Oba parametry pozwalają na ocenę ryzyka nawrotu choroby oraz wdrożenie odpowiedniego leczenia. To ostatnie jest szczególnie ważne w przypadku nowotworów wykazujących obecność mutacji genów kodujących białka związane z opornością na leki (MRP ang. multidrug resistance-associated protein). Komórki nowotworowe, w których potwierdzono ekspresję tych genów mają zdolność usuwania leków przeciwnowotworowych z wnętrza. Ponadto Real-time PCR jest wykorzystywany w badaniach dotyczących mikroRNA.
Podsumowanie
Metoda qPCR posiada olbrzymi potencjał diagnostyczny, ponieważ przy nieskomplikowanym procesie przedanalitycznym pozwala na uzyskanie precyzyjnych wyników. Jednak wprowadzenie tej techniki do rutynowych laboratoriów diagnostycznych wymaga opracowania jednolitych procedur obejmujących przygotowanie matrycy do badań, optymalizację warunków prowadzenia reakcji czy też analizy danych. Jedną z przeszkód jest również wciąż wysoki koszt aparatury i odczynników do qPCR.
Piśmiennictwo:
1. Wyczałkowska-Tomasik A. I wsp.: Łańuchowa reakcja polimerazy w czasie rzeczywistym – zastosowanie w badaniach naukowych i diagnostyce medycznej. Przegląd Lekarski 2009/66/4: 209-212.
2. Wiedro K., Stachowska E., Chlubek D.: Łańcuchowa reakcja polimerazy z analizą w czasie rzeczywistym (RT-PCR). Annales Academiae Medicae Stetinensis 2007, 53, 3, 5-9.
3. Studzińska A., Tyburski J., Daca P., Tretyn A.: PCR w czasie rzeczywistym. Istota metody i strategie monitorowania przebiegu reakcji. Biotechnologia 2008, 1 (80), 71-85.
4. Kubista M. et al: The real-time polymerse chain reaction. Molecular Aspects of Medicine 27 (2006) 95-125.
5. Valasek, Mark A., Joyce J. Repa. The power of real-time PCR. Adv Physiol Educ 29: 151–159, 2005;
6. VanGuilder H. D., Vrana K. E., Freeman W. M.: Twenty-five years of quantitative PCR for gene expression analysis. BioTechniques 44:619-626 (25th Anniversary Issue, April 2008).
7. Guest Editor’s Introduction: The ongoing evolution of qPCR. Methods 50 (2010), 215-216.
8. Kitchen R. R., Kubista M., Tichopad A.: Statistical aspects of quantitative real-time PCR experiment design. Methods 50 (2010), 231-236.
9. Real-Time PCR Applications Guide, Bio-Rad Laboratories, Inc., 2006.
10. Derveaux S., Vandesompele J., Hellemans J.: How to do successful gene expression analysis using real-time PCR. Methods 50 (2010), 227-230.
11. Mackay I. M., Arden K. E., Nitsche A.: Real-time PCR in virology. Nucleic Acids Research, 2002, Vol.30, No.6, 1292-1305.
12. Espy M. J. et al: Real-time PCR in Clinical Microbiology: Applications for routine laboratory testing. Clinical Microbiology Reviews, Jan. 2006, Vol. 19, No. 1, p. 165-256.
13. Mackay I. M.: Real-time PCR in microbiology laboratory. Clinical Microbiology and Infection, March 2004, Vol. 10, No. 3, 190-212.





